谢晓亮课题组发表Nature Genetics:高分辨率单细胞三维基因组测序新技术揭示多增强子中心的空间结构
2025年07月08日
在动物基因组中,增强子(enhancer)是一类重要的DNA顺式调控元件,能够跨越长距离“远程”调控基因在特定时间和空间中的表达。哺乳动物基因组中存在数量庞大的增强子,多个增强子常常协同调控一个基因的表达。然而,这些增强子在细胞核三维空间中如何组织、如何实现协同调控基因表达,目前仍不清楚。这背后的难题在于,现有的三维基因组测序方法要么只能获取细胞群体的平均结构,要么分辨率太低,难以观察单个细胞中精细的结构特征。
2025年7月2日,昌平实验室/北京大学生物医学前沿创新中心谢晓亮教授课题组在Nature Genetics在线发表了题为 “Single-cell Micro-C profiles 3D genome structures at high resolution and characterizes multi-enhancer hubs” 的研究论文。该研究首次开发出一种能在单细胞水平、千碱基级别精度下绘制基因组三维结构的新技术:scMicro-C(单细胞Micro-C),为研究精细染色质空间结构提供了强有力的工具。基于此技术,研究者在单细胞三维结构中观察到多个增强子同时靠近目标基因的启动子,形成一种新的空间结构:多增强子中心(multi-enhancer hub)。

与传统的单细胞Hi-C方法相比,单细胞Micro-C技术使用微球菌核酸酶(MNase)进行染色质切割,并结合优化的末端修复和连接策略,大幅提升了染色质接触捕获效率,使得研究者可以在单个细胞中可检测到近百万个染色质接触数目。利用该技术,研究人员成功绘制了人B淋巴母细胞的高分辨率染色质接触图谱,并重构了其5千碱基分辨率的三维基因组结构。
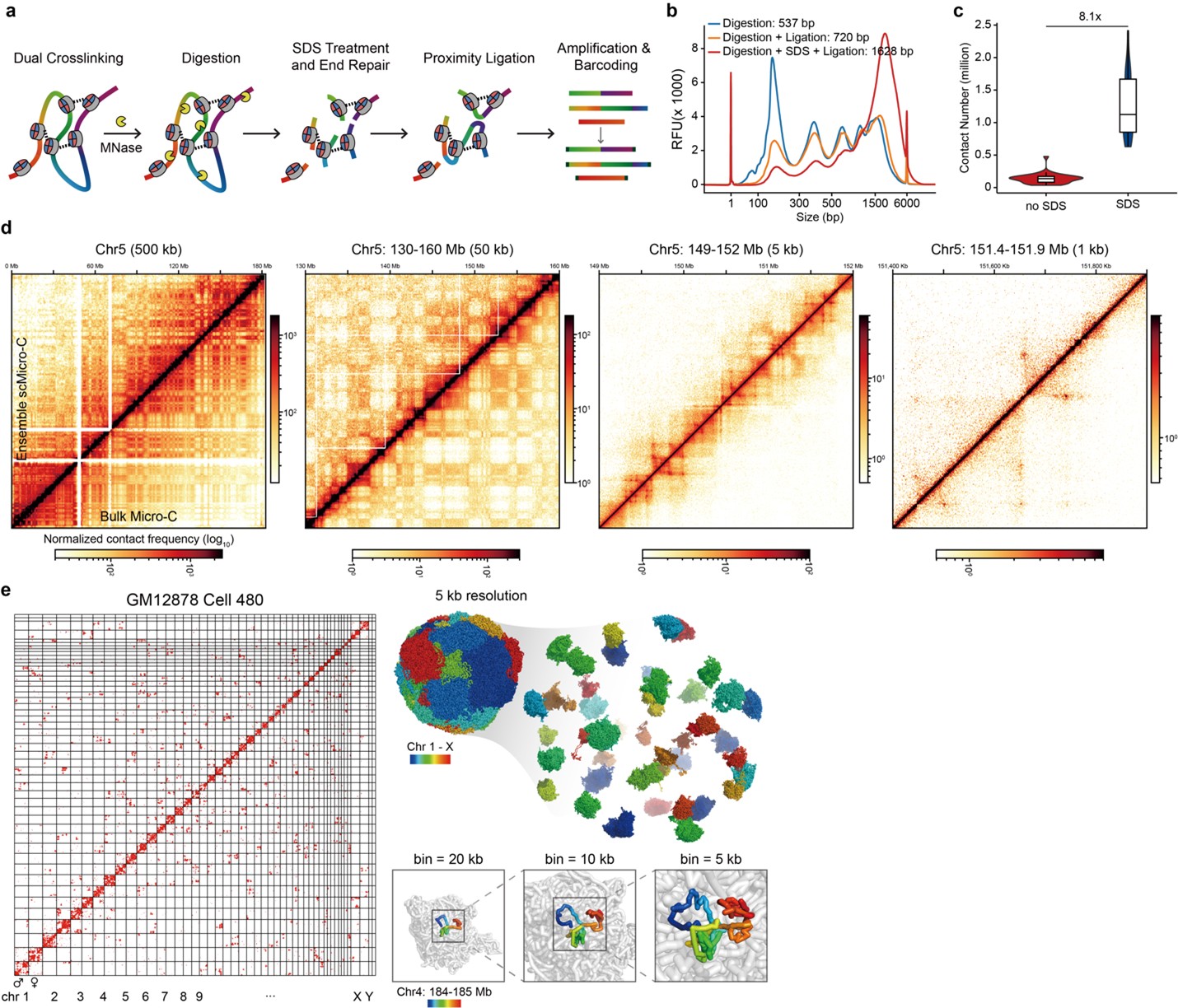
图1 | 单细胞Micro-C技术开发。
在对染色质互作图谱的深入分析中,研究人员发现了一种新的染色质结构: “启动子-增强子条纹(Promoter-Enhancer Stripe, PES)”。在二维互作图谱上。PES是一条锚定在启动子上、沿转录方向延伸的“条纹”,且PES相关基因通常具有更高的转录活性。PES基因的基因体区域富集活性增强子标记(H3K27ac),且常包含多个增强子调控元件,提示PES结构可能反映了启动子与多个增强子之间的动态互作关系。
进一步实验表明,PES结构依赖于染色质结构维持蛋白cohesin介导的“环挤出”机制,研究者据此提出了“cohesin辅助启动子扫描下游增强子”的模型,为理解启动子如何在空间中整合多个增强子信号提供了新机制。
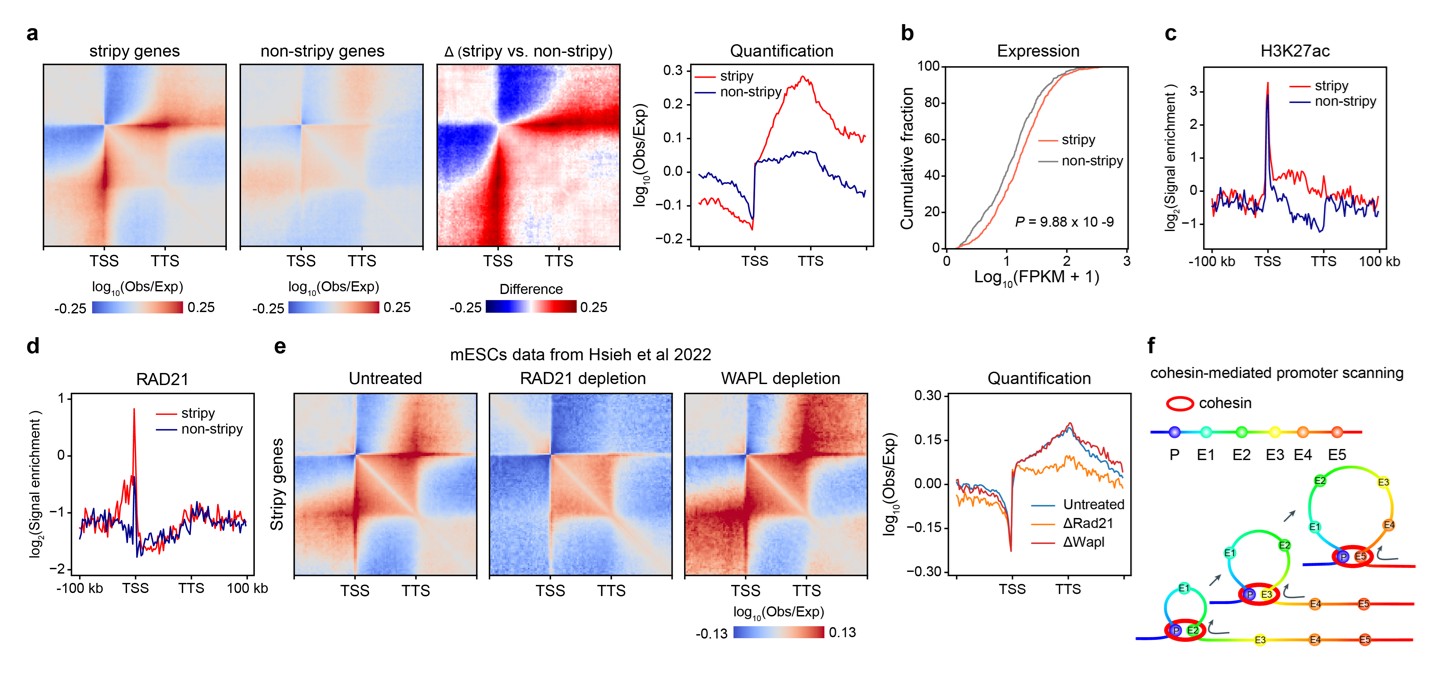 图2 | 启动子-增强子条纹结构的识别与功能分析。
图2 | 启动子-增强子条纹结构的识别与功能分析。
以关键免疫转录因子基因 EBF1 为例,研究者发现其基因体上的七个增强子之间存在丰富的互作,不仅与启动子发生联系,也彼此形成增强子-增强子互作。单细胞三维重建结果显示:这些增强子在空间中倾向于聚集成团,同时靠近目标基因启动子,即形成“多增强子中心”结构。这一现象在多个PES基因中广泛存在(如 IRF2、PIEZO2 等),说明多个增强子是“团队作战”而非“单打独斗”。
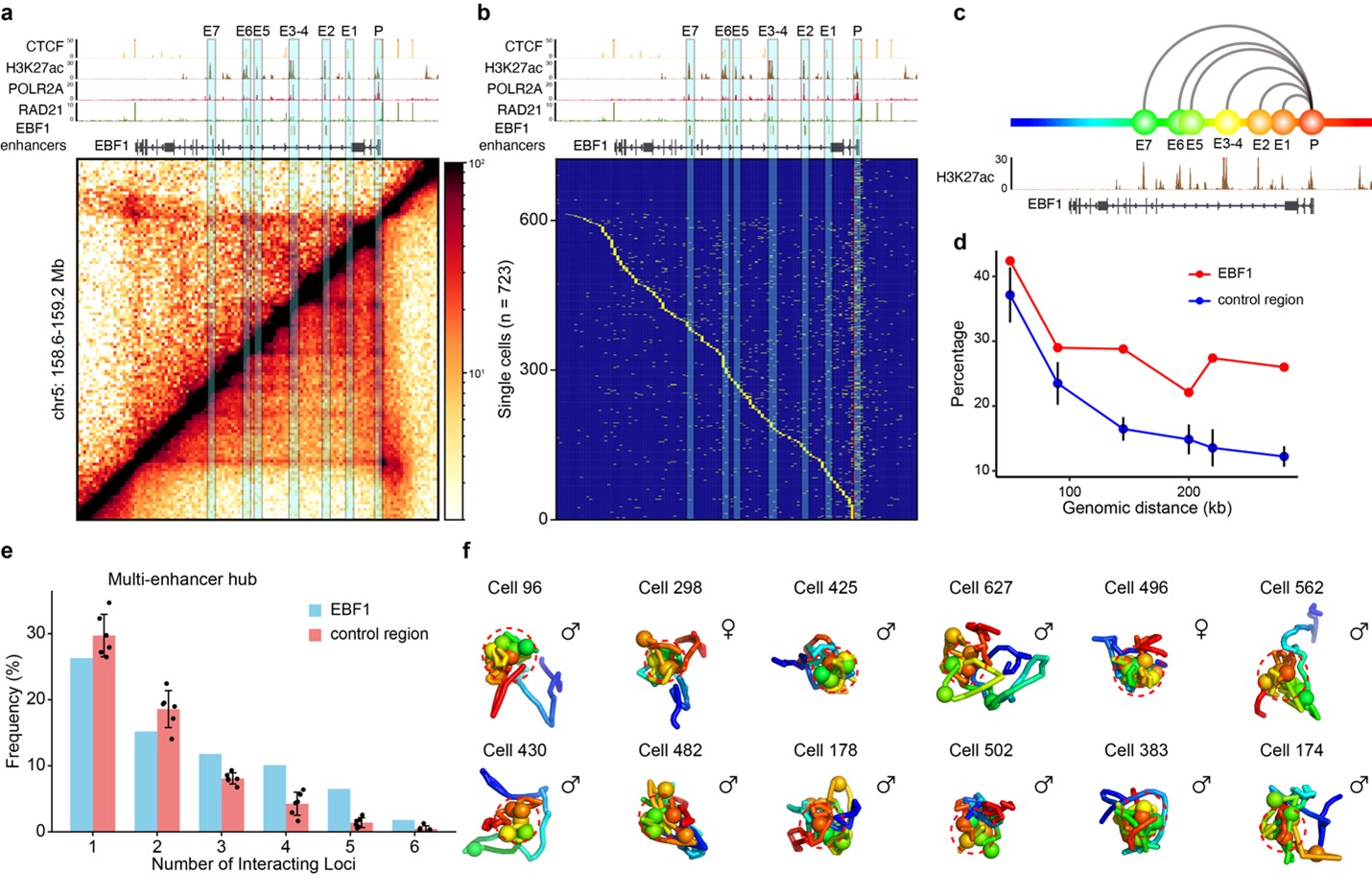
图3 |多增强子中心结构的发现与解析。
这项研究为我们认识基因调控提供了新的空间维度视角:首次在单细胞中捕捉增强子间的协同模式,揭示启动子如何在三维空间中“搜寻”增强子,提供高分辨率三维基因组结构研究的新工具与框架。
该研究由北京大学生物医学前沿创新中心/生命科学学院博士毕业生吴烘贵和张剑坤,以及斯坦福大学谭隆志教授为并列第一作者完成。昌平实验室/北京大学生物医学前沿创新中心谢晓亮教授为该论文通讯作者。该研究得到昌平实验室的支持和资助。
论文链接: