单细胞组学技术近年来蓬勃发展,已成为研究细胞功能及其背后的基因调控机制的重要手段,目前许多不同组学已能在单细胞精度下进行检测,包括转录组、染色质开放组、三维染色质组、DNA甲基化组等等,每种组学反应的是细胞状态的一个侧面,产生的数据需要进行整合分析才能更全面地刻画细胞内的基因调控状态,并深入揭示调控机制。虽然近年已有能在同一个单细胞内检测多种不同组学的实验方法被开发出来,但这类方法具有较高的技术门槛,且数据质量普遍低于单组学测序,更为重要的是,可同时检测的组学组合受到限制,在同一细胞内检测所有组学并不现实。
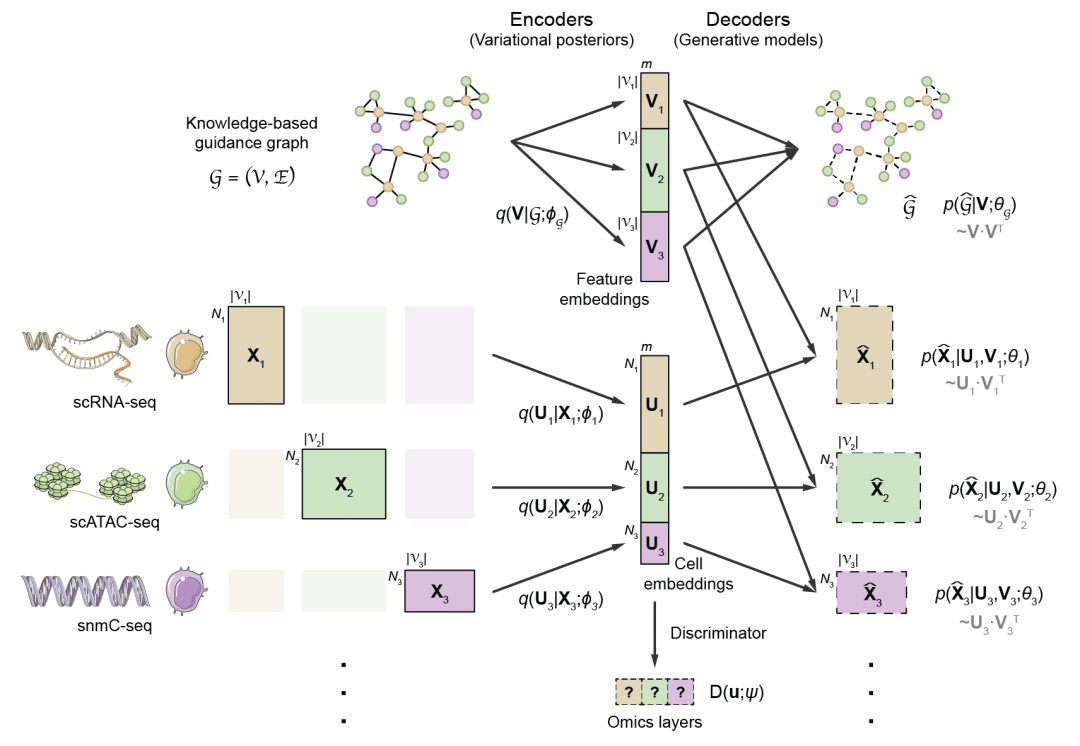
在这一大背景下,通过计算方法整合多个非配对的单组学数据集具有重要意义,一定程度上可以在无需特殊实验技术的情况下近似多组学实验,拓展单细胞多组学分析的边界。计算整合单细胞多组学数据的主要挑战在于不同组学具有不同的特征空间,例如转录组以基因为特征,而染色质开放组则以染色质开放区域为特征,不同特征空间的细胞之间缺乏可比性;与此同时,技术的发展使得单细胞测序的细胞通量迅速增长,现已达到百万细胞级别,如何有效整合如此大规模的多组学异质数据也是一个重要的计算挑战。
为了解决这一计算难题,实现高效、准确的单细胞多组学整合,2022年5月2日,北京大学生物医学前沿中心(BIOPIC)、北京未来基因诊断高精尖创新中心(ICG)、北京大学生命科学学院生物信息中心(CBI)、蛋白质与植物基因研究国家重点实验室的高歌课题组在期刊Nature Biotechnology上线上发表了题为“Multi-omics single-cell data integration and regulatory inference with graph-linked embedding”的研究论文,发布了基于深度学习模型的单细胞多组学数据整合与调控推断方法GLUE。